ORGANISATION COLLECTE TRAITEMENT PRESERVATION PARTAGE REUTILISATION
Partager les {méta}données
En général, le partage des données est d’abord interne, entre services différents d’une même structure, ou entre partenaires d’un projet. Une fois que les données d’un projet sont nettoyées et stabilisées, il est important de penser à les publier. Les données de la recherche peuvent être publiées via un dépôt disciplinaire, institutionnel ou plus généraliste tel que l’entrepôt national Recherche Data Gouv. Il est recommandé de publier ces jeux de données dans un entrepôt sécurisé générant automatiquement un DOI (Digital Object Identifier). Certaines restrictions à la diffusion des données peuvent exister, notamment dans le cas de données personnelles ou sensibles. Le plus important est de se poser les bonnes questions telles que : quelles données partager ou publier ? Comment ? Dans quel délai ? Quelle licence sera associée aux données ? etc. [Source, Université Paris-Saclay]
- Pourquoi standardiser par la métadonnée?
- Quelles sont les métadonnées minimales à partager?
- Focus sur l'outil développé par le PNDB : MetaShARK
- Où partager ses {méta}données?
- Data paper
- Partager les codes sources
- Identifiant péréenne
Pourquoi standardiser par la métadonnée?
Standardiser par la métadonnée permet :
- la description fine, l’inférence, l’identification et l'interopérabilité des données
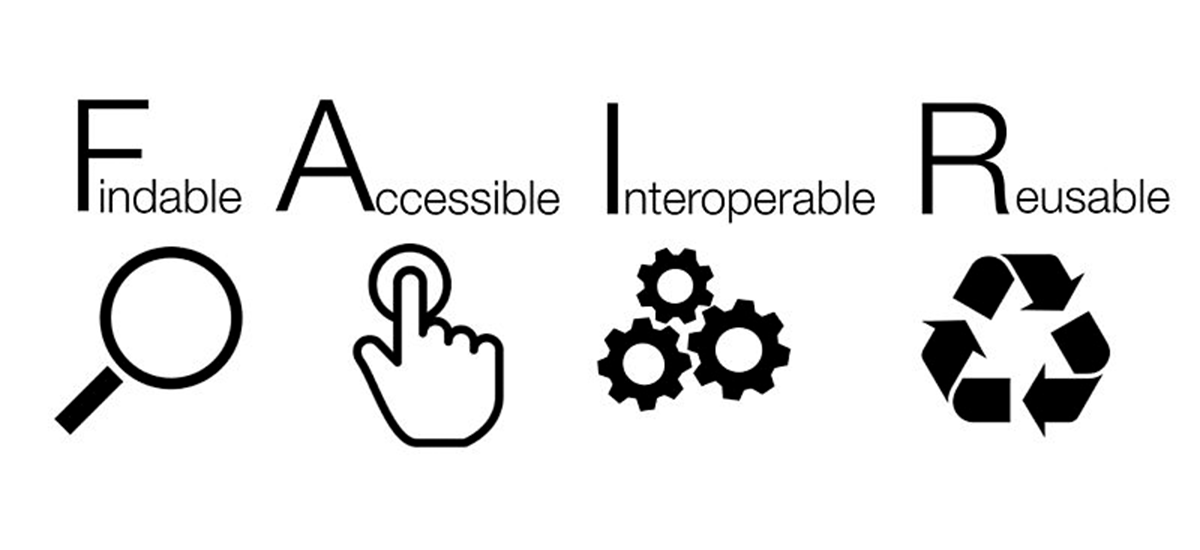
- s'intégrer dans l'approche la plus FAIR possible
- une meilleure reproductibilité en sciences écologiques.

L’Ecological Metadata Language - EML- est un standard pivot mondialement reconnu et qui a plus de 25 ans de retours d’expériences par les écologues / écoinformaticiens du national center for Ecological Analysis and Synthesis dans le cadre du projet DataONE
Exemple de cas pratique de standardisation par la métadonnée (à gauche) et "traduction en EML" (à droite)

Images et textes tirés de M.B. Jones et al., 2006 https://www.annualreviews.org/doi/10.1146/annurev.ecolsys.37.091305.110031
Quelles sont les métadonnées minimales à partager?
La gouvernance du PNDB, a validé une liste de métadonnées indispensables afin d'avoir un degrès de "FAIRitude" (cf. principes FAIR) minimal et relativement élévé
- Données ouvertes (CC-BY 4.0 compatible Etalab)
- Licence obligatoire
- Lien direct de téléchargement des jeux de données brutes
- Périmètre thématique (Toute la biodiversité y compris la paléo- et archéo-biodiversité)
- Périmètre géographique (Données produites par la France)
- Couverture temporelle (à minima une date d’acquisition de données)
- Résumé
- Titre, auteurs et contacts
- Cadre d’acquisition (a minima via un champ texte)
- DOI / identifiants uniques
- couverture taxonomique (si présence de taxons)
- mots clés en lien avec Thesaurus
- Attributs des données (Dictionnaire des attributs de données avec unités et descriptions)
- Annotation sémantique (Mots-clés et noms d’attributs, ressources utilisables illimitées)
Ces informations minimales sont donc communes à toutes les fiches de métadonnées présentes dans le catalogue du PNDB
=> Le PNDB peut vous aider à identifier les informations minimales à collecter et partager lors de la rédaction d'un PGD [voir pages étapes du cycle de vie des données Organisation & Collecte] et vous former sur les principes FAIR et sur le cycle de vie des données [voir page Formations]
Focus sur l'outil développé par le PNDB : MetaShARK
Nota Bene : MetaShARK (pour Metadata Shiny Application for Resources and Knowledge) est un outil R&D financé par le Fond National pour la Science Ouverte, dans le cadre du projet OpenMetaPaper . Cet outil n'est pas disponible car actuellement en crash test interne
Il a pour objectif d'extraire et générer, avec le plus d’inférence/automatisation/suggestion possible, les métadonnées, c’est-à-dire les informations décrivant le contexte des données, et ce dans le format pivot international qui est EML.
MetaShARK est donc une application R Shiny en cours de développement sous forme d'une interface graphique et utilisant les packages R EML et EML Assembly Line. Cette interface permettra in fine de parcourir facilement les spécifications de l’EML et de saisir des métadonnées.
MetaShARK permettra notamment de :
- faire des liens entre des personnes, des projets, des objets (données, code source, protocoles, ...), des thèmes de recherche
- intégrer et générer des métadonnées liées à d'autres systèmes d'informations, entrepôts institutionnels
- générer V0 de data paper
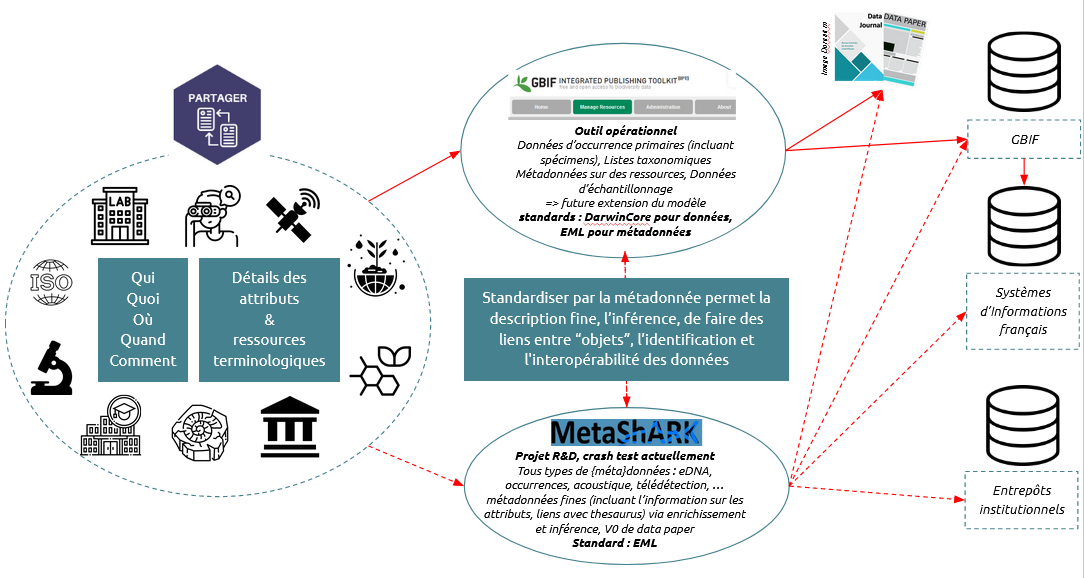
Nota Bene : toutes les {méta}données recherche moissonnées par le PNDB seront remontées au GBIF si ces dernières sont compatibles au format et standard "DarwinCore" (ex. Données d’occurrence primaires (incluant spécimens), Listes taxonomiques Métadonnées sur des ressources, Données d’échantillonnage).
Où partager ses {méta}données?
Dans le cadre de la politique de Science ouverte portée par le Ministère de l'Esignement Supérieur et de la Recherche, le stockage de vos données doit se faire dans les entrepôts de données de votre institution ou entrepôt institutionnel. nous pouvons notamment citer les entrepôts INRORES du CNRS-INEE , dataverse de l'IRD , dataverse du CIRAD, ...
[Voir cette liste pour le domaine de la biodiversité]
=> Un groupe de travail coordonnée par le PNDB et le GBIF France, au sein de l'unité d'Appui et de Recherche - PatriNat - a rédigé une note synthèse "comprendre, partager, réutiliser les données de biodievrsité, complémentarité des systèmes d'information GBIF, SIB, SINP, PNDB". Cette dernière a pour objectif d'accompagner lescommunautés dans la compréhension, le partage et l'utilisation des données de biodiversité, et donc dans la préservation des {méta}données.
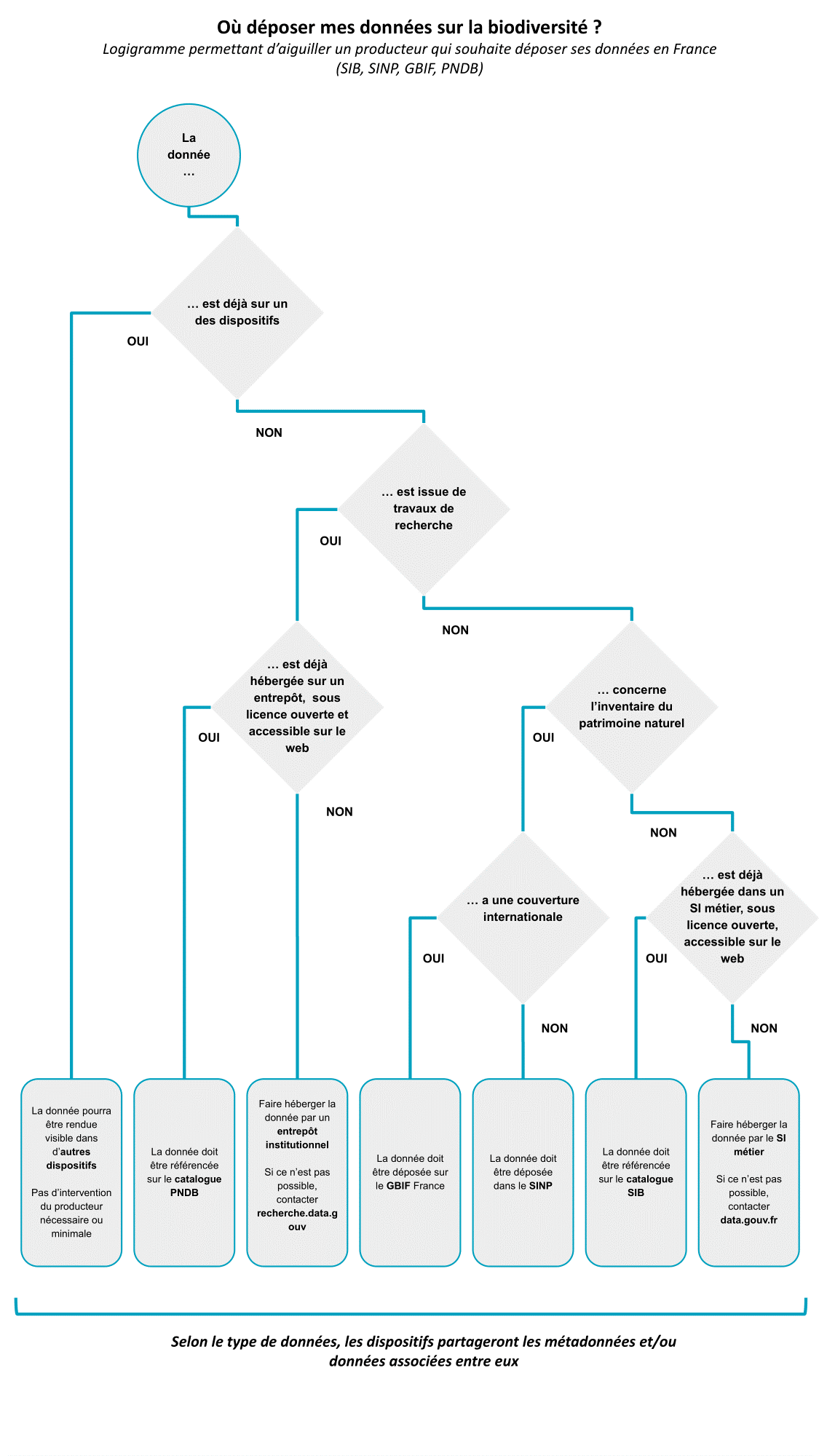
De plus Recherche Data Gouv propose un logigramme pour savoir "où déposer ses données?" voir le logigramme
Data Paper
Un data paper est une publication scientifique qui décrit précisément un jeu de données, et informe la communauté scientifique de son existence, de ses modalités et de son potentiel de réutilisation.
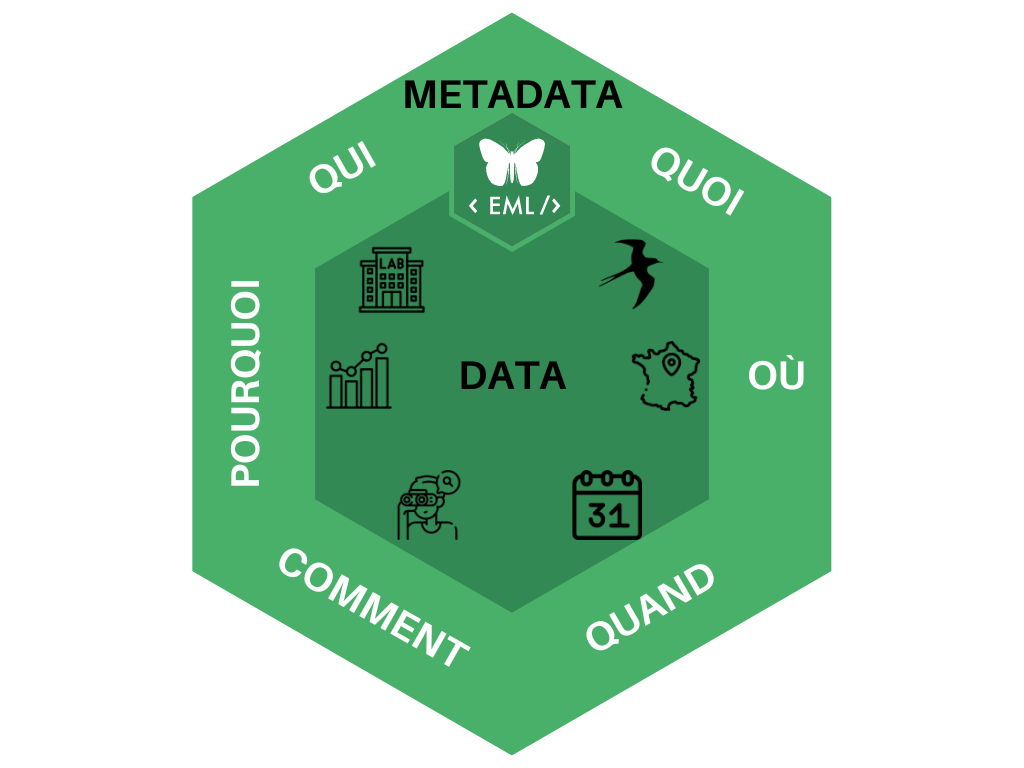
=> le qui, quoi, où, quand, commet, pourquoi des données = métadonnées
- Il décrit un jeu de données via les métadonnées
- Il démontre la qualité et quantité des données, leur méthode de collecte et de traitement.
- Il montre l’originalité et la portée de ce jeu de données, ainsi que leur potentiel pour des utilisations futures (arguments décisifs pour l’acceptation de la publication).
- Il donne généralement accès au jeu de données, dans un fichier attaché ou par un lien pérenne (URL, DOI) vers l’entrepôt (data repository, ou repository of research data) où le jeu est déposé. Le jeu de données décrit dans le data paper est en effet normalement accessible gratuitement à tous pour une réutilisation pouvant être commerciale (en France, licence ouverte Etalab V2.0 compatible licence CC-BY 4.0). Toutefois, selon les possibilités offertes par les entrepôts, les données peuvent être temporairement sous embargo, ou accessibles uniquement sur demande.
Source IRD : https://data.ird.fr/datapapers/ et adaptée
Cette valorisation de Data paper se fait en lien avec le GBIF France et dans le cadre du projet OpenMetaPaper (soutenu par le Fond National pour la Science Ouverte) qui vise à "booster" la plublication de data papers et de décrire finement les métadonnées
Pour aller plus loin autour des data papers : voir la documentation dédiée du service Information Scientifique et Technique du CIRAD
Partager les codes sources
L'approche partage de codes sources sur GitHub / GitLab, puis dans le but de proposer un haut degré de FAIRitude, les conseils sont de définir les dépendances via conda, puis de les mettre à disposition via des containers (par exemple via Docker ou Singularity), puis le cas échéant d'en faire un outil Galaxy-Ecology pour un accès direct aux fonctions du code source.
| ACCES A LA PLATEFORME D'ANALAYSE GALAXY-ECOLOGY |
=> Le PNDB peut vous accompagner dans la découverte de Galaxy-E (voir tutoriels en écologie) et/ou vous inititer à cette plateforme (voir Formations)
Identifiant péréenne
Un identifiant pérenne (Persistent identifier ou PID) est un code alphanumérique associé à un objet ou ressource de façon permanente. Il est disponible et gérable à long terme ; il ne changera pas si l’objet est renommé ou déplacé (changement de site, d’entrepôts de données…).Il peut s'agir par exemple d'un DOI, ou encore d'un numéro ORCID. [Source OPIDoR].
Lors que vous déposez vos données et métadonnées dans un entrepôts thématiques et/ou institutionnels, ce dernier vous attribue un DOI.
=> Le PNDB peut vous accompagner dans le choix du bon entrepôt de données, et relaie par son catalogue votre jeux de données et ses métadonnées via le DOI attribué par l'entrepôts.
Pour aller plus loin :
ORGANISATION COLLECTE TRAITEMENT PRESERVATION PARTAGE REUTILISATION