OpenMetaPaper
Résumé
« La nature décline globalement à un rythme sans précédent dans l’histoire humaine – et le taux d’extinction des espèces s’accélère, provoquant dès à présent des effets graves sur les populations humaines du monde entier ». Afin de mieux protéger nos écosystèmes, nous devons mieux les connaître, et pour ce faire, faciliter l’accès et le partage des informations générées et utilisées par les chercheurs pour construire la connaissance scientifique en commençant par l’ouverture la plus généralisée possible des données. Dans les disciplines scientifiques liées à la donnée de biodiversité, la culture du partage de la donnée n’est pas aussi ancrée que dans les disciplines de la biologie moléculaire par exemple. Pour autant, l’ouverture des données n’est pas suffisante et il est primordial d’y associer des métadonnées détaillées en utilisant des standards largement utilisés à l’international. Dans ce cadre, il apparaît que le domaine de l’écologie possède un avantage face à la multiplicité des standards existants au sein des communautés liées aux technologies *omiques : l’existence d’un langage de métadonnée dédié, modulaire et exhaustif, l’EML. Le projet OpenMetaPaper propose de compléter les initiatives existantes au niveau du Pôle national de données de biodiversité, et en lien avec notamment le GBIF et le projet GO FAIR BiodiFAIRse afin de booster l’ouverture des données de recherche en écologie et d’accélérer les initiatives en cours autour de l’utilisation du standard EML et de ses liens avec les autres standards de données et métadonnées en écologie et dans les disciplines proches. Particulièrement, le projet propose de mettre l’accent sur la publication scientifique, objet de recherche principal de la valorisation des activités scientifiques, en testant un dispositif permettant de :
- booster la production de “data paper” par la communauté en écologie
- augmenter l’impact de ces articles en facilitant la publication de tel matériel dans des revues à haut facteur.



Fonctionnement
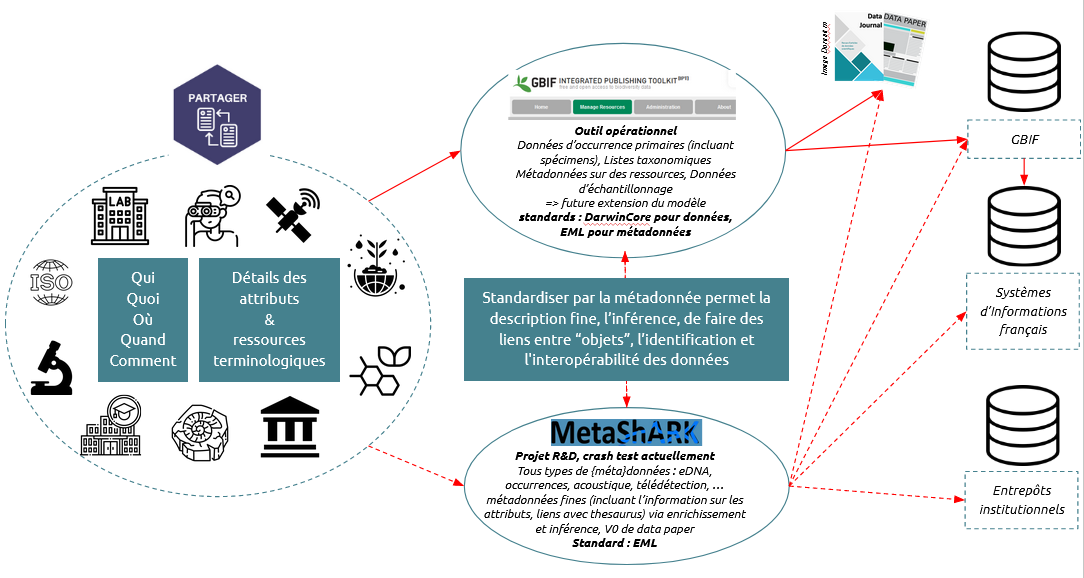
Nota Bene : MetaShARK (pour Metadata Shiny Application for Resources and Knowledge) est un outil R&D financé par le Fond National pour la Science Ouverte, dans le cadre du projet OpenMetaPaper . Cet outil n'est pas disponible car actuellement en crash test interne
Data paper
Un data paper est une publication scientifique qui décrit précisément un jeu de données, et informe la communauté scientifique de son existence, de ses modalités et de son potentiel de réutilisation.
=> le qui, quoi, où, quand, commet, pourquoi des données = métadonnées
- Il décrit un jeu de données via les métadonnées
- Il démontre la qualité et quantité des données, leur méthode de collecte et de traitement.
- Il montre l’originalité et la portée de ce jeu de données, ainsi que leur potentiel pour des utilisations futures (arguments décisifs pour l’acceptation de la publication).
- Il donne généralement accès au jeu de données, dans un fichier attaché ou par un lien pérenne (URL, DOI) vers l’entrepôt (data repository, ou repository of research data) où le jeu est déposé. Le jeu de données décrit dans le data paper est en effet normalement accessible gratuitement à tous pour une réutilisation pouvant être commerciale (en France, licence ouverte Etalab V2.0 compatible licence CC-BY 4.0). Toutefois, selon les possibilités offertes par les entrepôts, les données peuvent être temporairement sous embargo, ou accessibles uniquement sur demande.
Source IRD : https://data.ird.fr/datapapers/ et adaptée
Cette valorisation de Data paper se fait en lien avec le GBIF France et dans le cadre du projet OpenMetaPaper (soutenu par le Fond National pour la Science Ouverte) qui vise à "booster" la plublication de data papers et de décrire finement les métadonnées
Pour aller plus loin autour des data papers : voir la documentation dédiée du service Information Scientifique et Technique du CIRAD
Financement
Ce projet est possible avec le concours financier du Fond National de la Science Ouverte (FNSO), dans le cadre de la politique générale "Science ouverte" du Ministère de l'Enseignement supérieur et de la Recherche / numéro de projet : AAPFNSO2019OpenMetaPaper-14026

